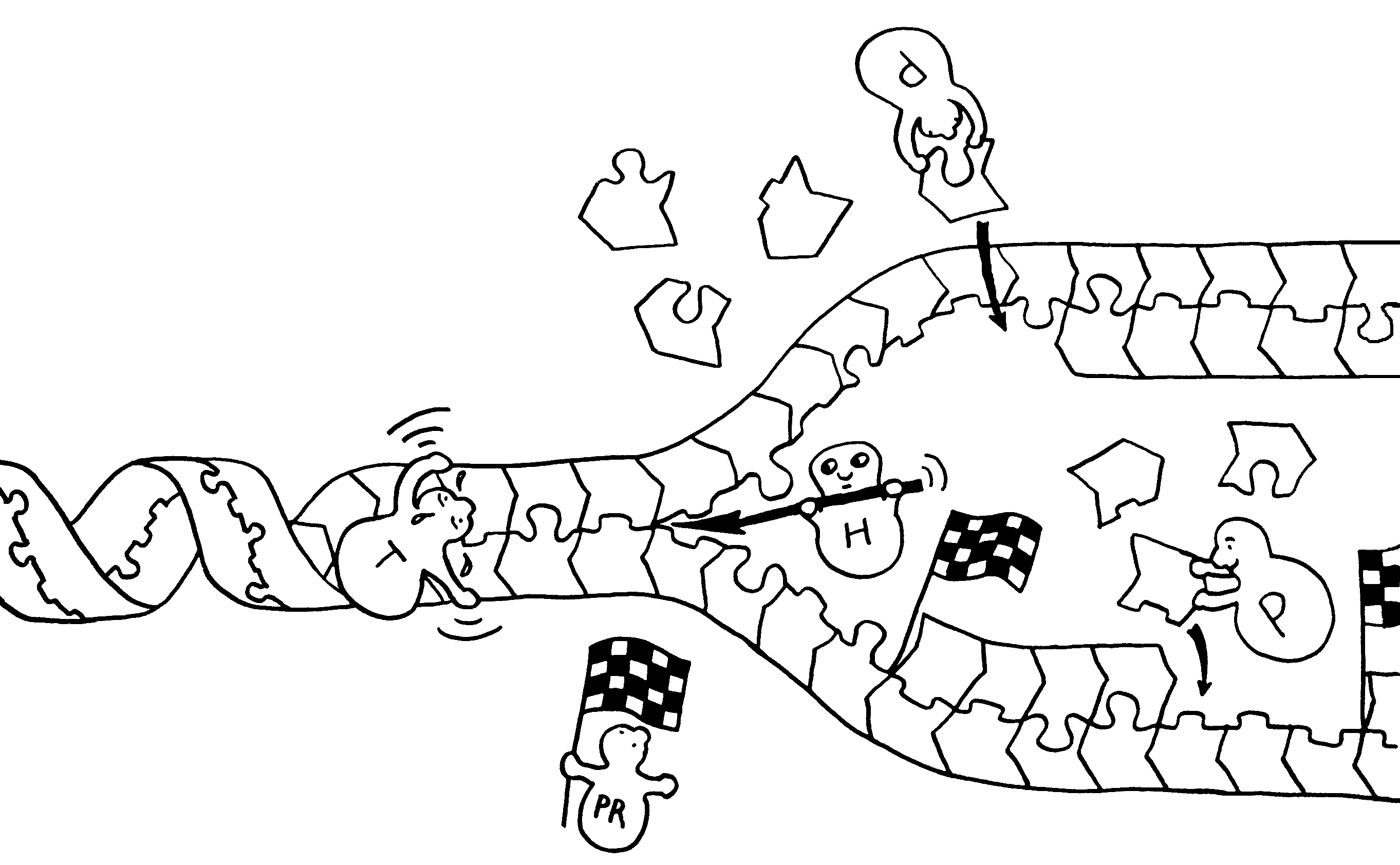
An einer sogenannten Replikationsgabel entstehen aus einem DNA-Doppelstrang zwei, die genau dieselben Erbinformationen enthalten: Die Abfolge der Puzzleteile – Nukleotide genannt – bleibt gleich. Jedes Nukleotid besteht aus einem Zucker, einer Phosphatgruppe und einer Base (s. o.). An der Verdopplung sind zahlreiche Enzyme beteiligt: Die Topoisomerase (T) windet die Doppelhelix auseinander, damit der Doppelstrang zugänglich wird. Die Helikase (H) mach aus dem Doppelstrang zwei Einzelstränge. Die Polymerasen (P) ergänzen jedes Nukleotid um das passende Gegenstück. Dabei wachsen beide neuen Einzelstränge in 5′-zu-3′-Richtung: in Richtung der Puzzleteil-Pfeile. Die Gabelung verschiebt sich allmählich nach links. Der obere der beiden neuen DNA-Stränge wächst einfach in dieselbe Richtung. Der untere neue Strang muss von links nach rechts wachsen, also notgedrungen stückchenweise. Dazu stellt die Primase
(PR) in bestimmten Abständen sogenannte Primer bereit, an denen jeweils ein neues Fragment beginnt. Wenn ein Fragment an den vorhergehenden Primer stößt, werden die Stücke verbunden. Weiter rechts (nicht im Bild) winden sich die beiden neuen Doppelstränge wieder zu Doppelhelices.
Sie dürfen diese Zeichnung gerne in Folien etc. übernehmen, sofern Sie die Quelle angeben: Dr. Andrea Kamphuis, https://autoimmunbuch.de