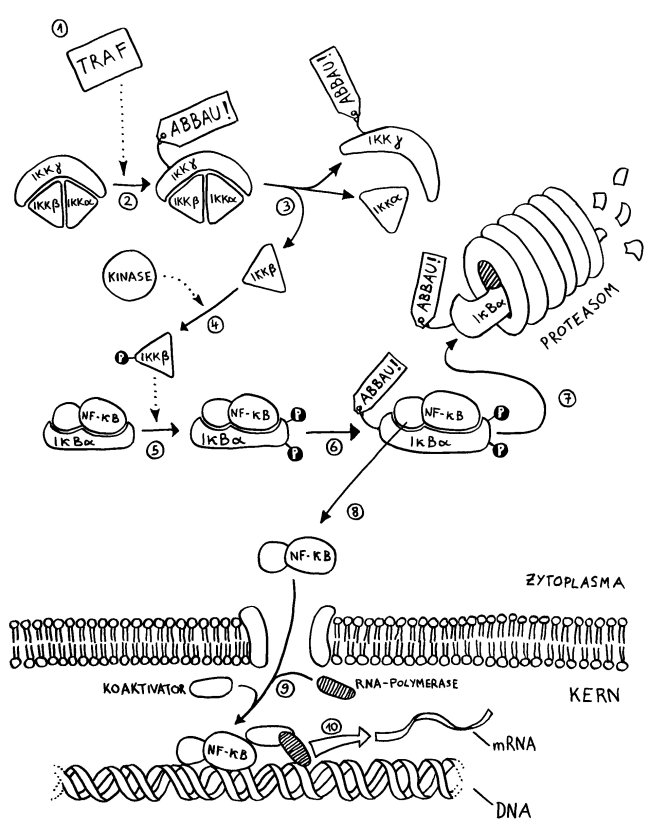
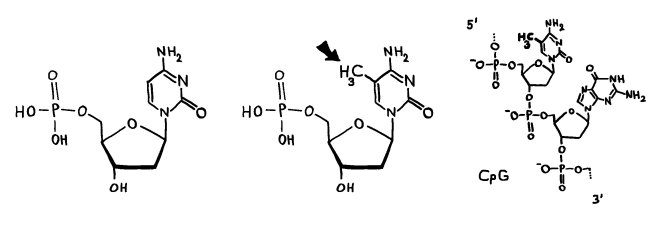
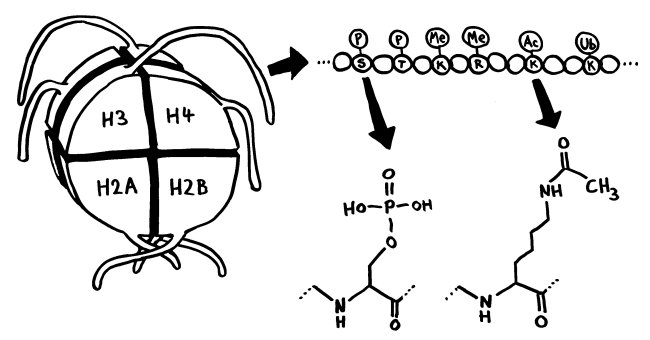
So dröges Zeug muss halt auch mal sein: einer der wichtigsten entzündungsfördernden Signalwege in vielen Immunzellen; nähere Erläuterungen folgen im Buch.
1 Ein TRAF (TNF-Rezeptor-assoziierter Faktor) wurde an der Innenseite eines Rezeptors in der Zellmembran aktiviert und löst den weiteren Signalweg aus.
2 Er ubiquitiniert IKKγ im IKK-Komplex, der daraufhin seine Gestalt ändert. (Bei der Ubiquitinierung wird einem Protein das Protein Ubiquitin angeheftet, wodurch es zum Abbau freigegeben wird.)
3 Die Kinase IKKβ löst sich von IKKα und IKKγ.
4 IKKβ kann nun durch eine andere Kinase phosphoryliert und damit aktiviert werden.
5 Der Transkriptionsfaktor NF-κB (ein Heterodimer) ist noch an den Inhibitor IκBα gebunden. Aber jetzt phosphoryliert die aktivierte Kinase IKKβ den Inhibitor doppelt, …
6 … woraufhin er auch noch ubiquitiniert und damit zum Abbau freigegeben wird.
7 Der Komplex zerfällt: Der Inhibitor wandert in ein Proteasom und wird zerlegt.
8 Der Transkriptionsfaktor NF-κB ist befreit und kann durch eine Kernpore in den Zellkern einwandern.
9 Im Zellkern lagern sich ein Koaktivator und das Enzym RNA-Polymerase mit dem Transkriptionsfaktor zusammen.
10 Der Komplex dockt am Promotor des abzulesenden Gens an und startet dessen Transkription, also die Erzeugung einer Messenger- oder mRNA, die die Informationen für die Synthese eines Proteins (z. B. eines entzündungsfördernden Zytokins) enthält.
Die mRNA wandert anschließend ins Zytoplasma, wo sie als Vorlage für die Proteinsynthese (Translation) dient.
Vorlagen: Wikipedia und Abbas, 7. Auflage, Abb. 7.26