Die nächste Skizze fürs Buch:
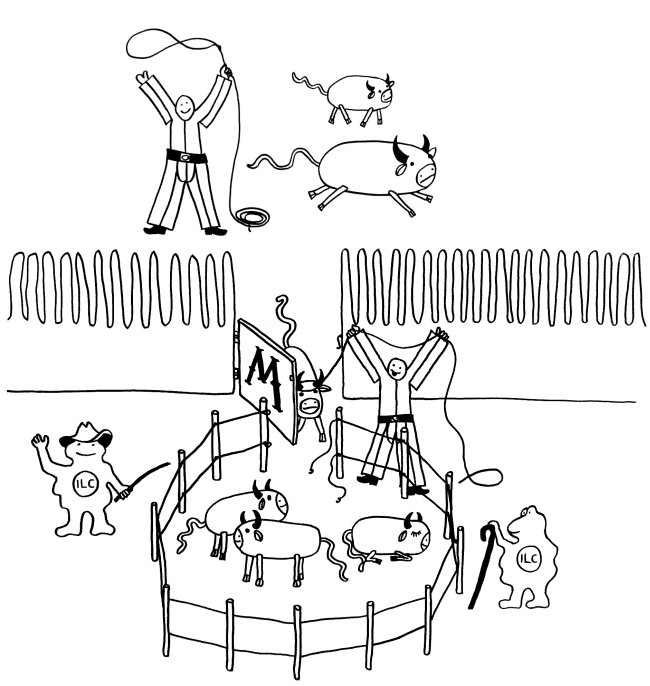 Eine der seltsamsten Entdeckungen, die in den letzten Jahren in unserem Darm gemacht wurden, ist die „Viehwirtschaft“ in den Peyer-Plaques – jenen Lymphfollikelhaufen unter dem Dünndarmepithel, in denen die lokale Immunabwehr organisiert wird.
Eine der seltsamsten Entdeckungen, die in den letzten Jahren in unserem Darm gemacht wurden, ist die „Viehwirtschaft“ in den Peyer-Plaques – jenen Lymphfollikelhaufen unter dem Dünndarmepithel, in denen die lokale Immunabwehr organisiert wird.
Das Epithel, hier als Palisade dargestellt, besteht aus einer einzelnen Schicht von Enterozyten, die seitlich durch tight junctions so eng miteinander verbunden sind, dass sich nichts Gefährliches zwischen ihnen hindurch aus dem Darmlumen (oben) in das darunter liegende Gewebe (unten) quetschen kann. Aber direkt über jedem Peyer-Plaque sitzt eine M-Zelle (die Pforte), die Substanzen aus der Nahrung, Bakterien und Bakterienbruchstücke aus dem Darmlumen aufnimmt und an ihrer Unterseite wieder ausscheidet (sog. Transzytose, von trans = hindurch und Endozytose).
Dort nehmen normalerweise dendritische Zellen, die unterhalb des Epithels Wache schieben, die Antigene auf, um sie zu verarbeiten und dann in den Peyer-Plaques den naiven T- und B-Zellen zu präsentieren. Dort werden die zu den Antigenen passenden Zellen der erworbenen Abwehr aktiviert; sie vermehren sich, wandern durch die Blutbahn und kehren in den Darm zurück, wo sie z. B. Infektionen bekämpfen.
Solange keine akute Infektion vorliegt, sondern den B-Zellen vor allem Antigene aus harmlosen Darmbakterien (Kommensalen) präsentiert werden, produzieren sie sogenanntes sekretorisches Immunglobulin A (sIgA): Dimere aus zwei Y-förmigen IgA-Antikörpern, die an ihren „Stielenden“ zusammengeheftet sind. Dieses sIgA wird durch das Epithel ins Darmlumen ausgeschieden, wo es an Antigene an der Oberfläche von Kommensalen bindet. sIgA löst keine Komplementreaktion und damit keine Entzündung aus, sondern hindert die Kommensalen einfach nur daran, sich an das Epithel anzulagern und damit die Enterozyten und die Immunzellen in der Nähe „nervös zu machen“.
Manchmal aber geschieht Seltsames: Das sIgA bindet an lebende Bakterien der Gattung Alcaligenes, klopft dann gewissermaßen an die M-Zellen-Tür und wird mitsamt der Bakterien durch die M-Zellen in die darunter liegenden Peyer-Plaques eingeschleust. Ich habe das sIgA hier als Cowboys und die Alcaligenes-Zellen als Rinder dargestellt. Im Peyer-Plaque – dem Corral – werden die Bakterien nicht etwa abgetötet, sondern sie leben weiter, werden mit Nährstoffen versorgt und vermehren sich offenbar sogar.
Noch ist nicht ganz auszuschließen, dass es sich um eine perfide Ausbeutung durch die Bakterien handelt. Wahrscheinlicher ist diese Form der „Viehhaltung“ aber eine Symbiose: Alcaligenes lässt sich in geschützter Umgebung versorgen und stimmt dafür das lokale Immunsystem tolerant, indem es ihm ständig kommensalentypische Antigene präsentiert. So werden gefährliche Überreaktionen auf harmlose Kommensalen vermieden.
Wichtig ist, dass die Bakterien nicht aus ihrem Corral ausbrechen. Dafür sorgen offenbar ILCs – die vor wenigen Jahren entdeckten innate lymphoid cells, die an der Grenze zwischen angeborenem und erworbenem Immunsystem stehen. Rings um die Peyer-Plaques machen sie sich als Hirten verdient. Fehlen sie oder haben sie einen Defekt, so brechen die Alcaligenes-Bakterien aus, die örtlichen B-Zellen schalten auf die Produktion von Antikörpern des Typs IgG um, und der Dünndarm entzündet sich.
Quellen:
- Kunisawa J. und H. Kiyono (2012): Alcaligenes is Commensal Bacteria Habituating in the Gut-Associated Lymphoid Tissue for the Regulation of Intestinal IgA Responses. Front Immunol. 2012; 3: 65, doi: 10.3389/fimmu.2012.00065
- Sonnenberg G. F. et al (2012): Innate Lymphoid Cells Promote Anatomical Containment of Lymphoid-Resident Commensal Bacteria. Science 336: 6086; doi: 10.1126/science.1222551